近日,我校与中国农业科学院深圳农业基因组研究所联合培养硕士研究生杨龙波在《Nature Communications》上在线发表了题为"GWAS meta-analysis using a graph-based pan-genome enhanced gene mining efficiency for agronomic traits in rice"的研究论文。

丰富的水稻种质资源中积累了大量可以用来提高产量性状的优异自然变异。产量性状是复杂变异控制的复杂性状。目前对种质资源遗传变异尤其是复杂变异的解析尚不够深入,这一现状限制了产量性状相关基因的深入挖掘,进而影响了其在育种中的应用。
为了进一步克服传统GWAS方法对群体结构和样本量的敏感性,以及在低频变异分析和微效基因挖掘方面的局限性,进而整合更多大规模数据资源,实现对超大规模群体的高效基因挖掘。该研究采用meta-GWAS策略,整合来自6个不同种质资源群体的7765份水稻种质资源基因型和表型数据,挖掘产量相关新基因。
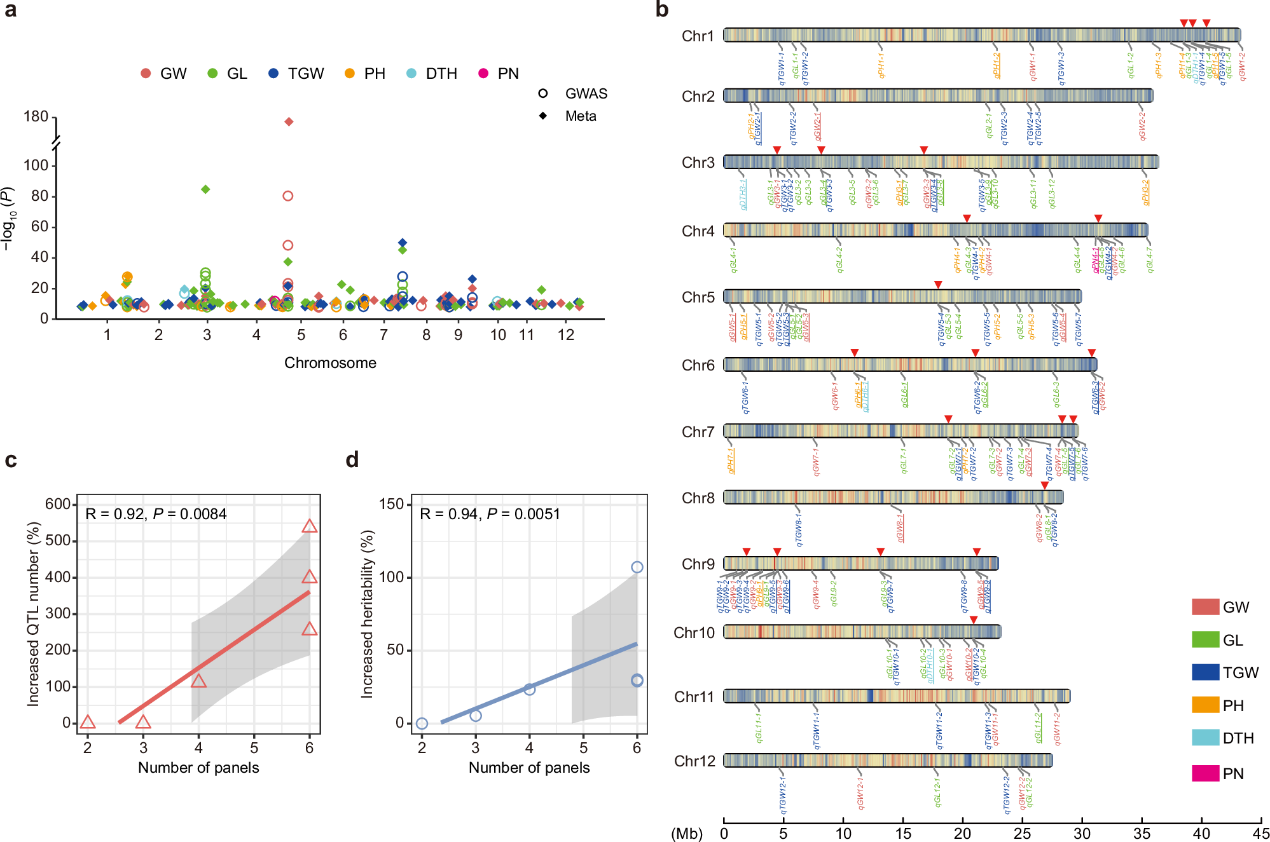
图1 通过六个水稻群体的meta-GWAS分析检测到的与性状相关的变异统计
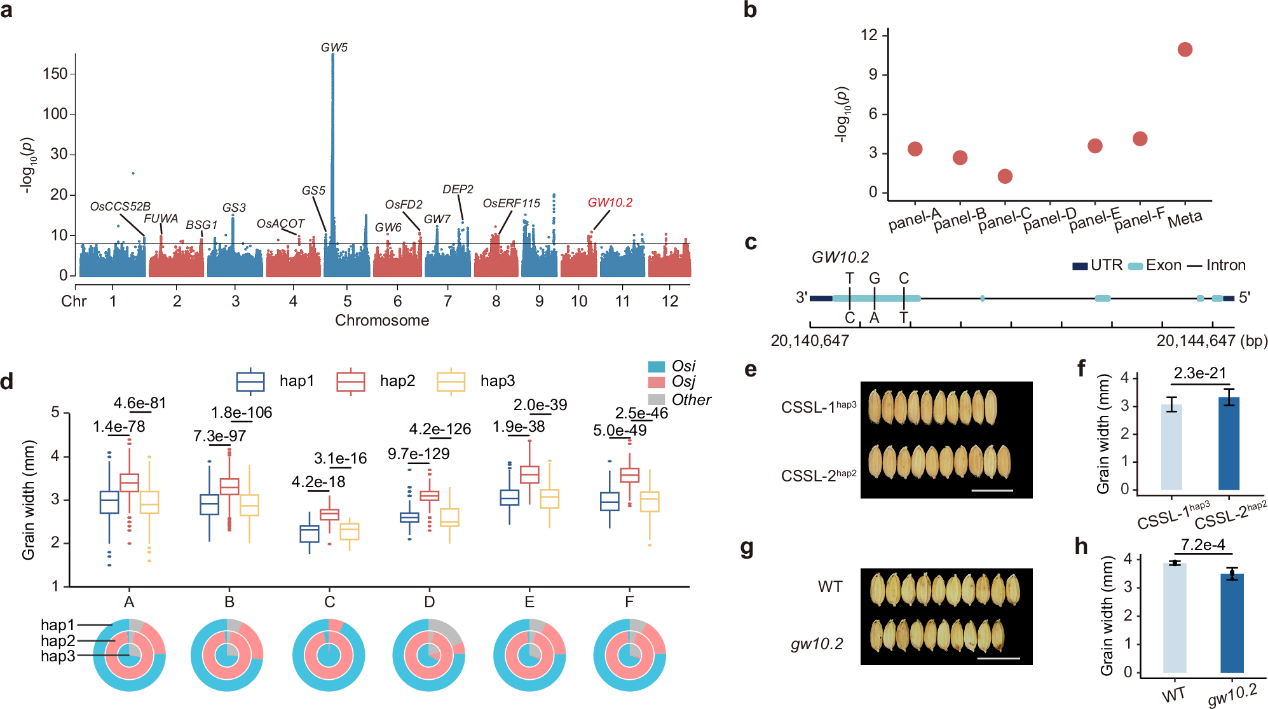
图2 粒宽的meta-GWAS及其功能分析
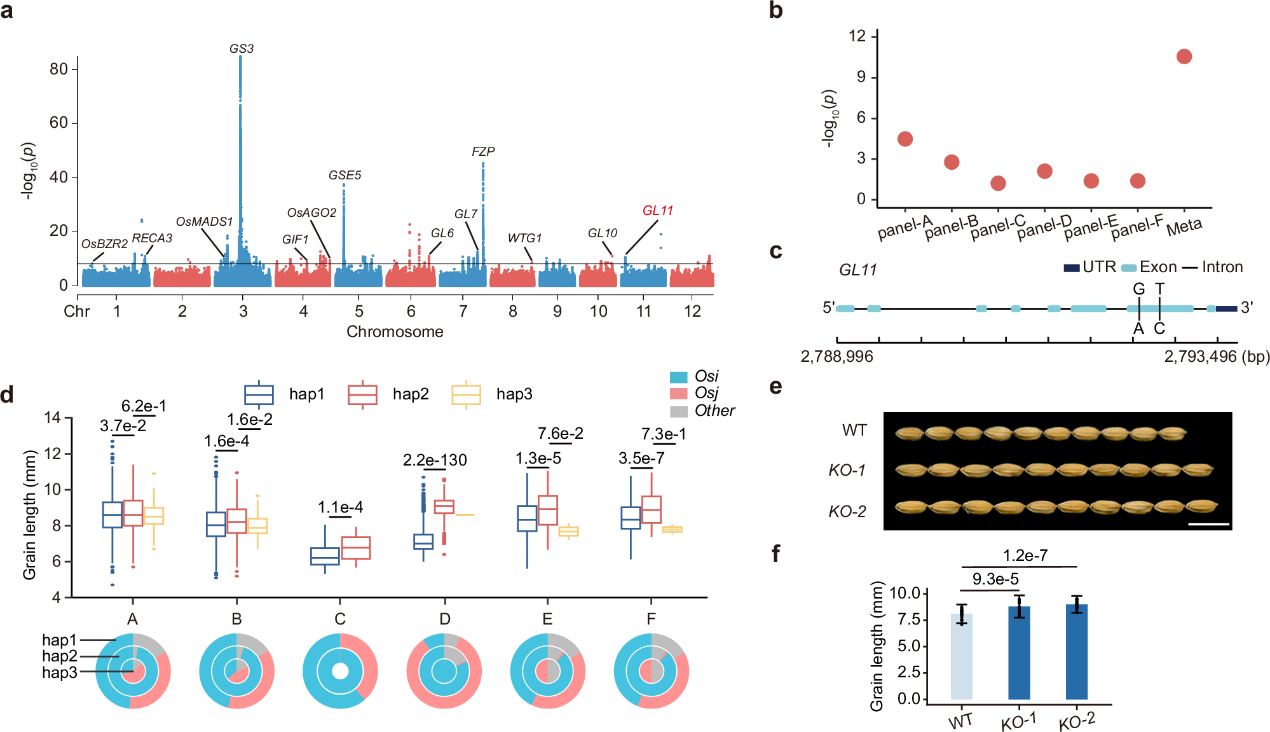
图3 粒长的meta-GWAS及其功能分析
基于图形泛基因组,共鉴定了6,604,898个单核苷酸多态性和42,879个结构变异。通过对6个群体开展独立GWAS分析,并进一步整合进行meta分析,共鉴定出156个与关键农艺性状相关的遗传位点,其中116个仅能通过meta-GWAS鉴定得到,显著提高了QTL的检测能力和遗传力的解释率。在此基础上,成功挖掘了水稻粒宽和粒长性状相关的新基因GW10.2和GL11,并通过分子遗传学实验进行了功能验证。该研究为水稻种质资源的高效深入挖掘和遗传改良提供了新的思路和工具,也为水稻高产分子设计育种提供了宝贵的基因资源。
我校与中国农业科学院深圳农业基因组研究所联合培养硕士研究生杨龙波、基因组所贺文闯副研究员、基因组所朱义旺副研究员(现福建省农科院)和基因组所博士生吕阳(现浙江省农科院)为论文共同第一作者。我校外聘硕士生导师、中国农业科学院深圳农业基因组研究所商连光研究员和崖州湾国家实验室钱前院士为论文的共同通讯作者,我校校内导师贾举庆老师为论文的共同作者。该研究得到国家自然科学基金基础科学中心、中国农业科学院科技创新工程科学中心和中国农科院青年创新专项资金资助。该工作得到了基因组所、中国水稻研究所和崖州湾科技城超级计算平台的支持。
原文链接:https://www.nature.com/articles/s41467-025-58081-1